Por: Cecilia Di Prinzio
(Acercaciencia.com)
Tan solo ha transcurrido un año desde que la OMS declaró la pandemia de enfermedad por coronavirus (COVID-19) y, sin dudas, en muchas partes del mundo estamos agotados. Sin embargo, un año resulta insignificante si consideramos escalas de tiempo evolutivo. Algo similar ocurre si tenemos en cuenta a todos los virus que han infectado a los seres vivos a lo largo de millones de años. Pero no podemos obviar que preguntas recurrentes como ¿hasta cuándo estará entre nosotros este virus? ¿cuánto durará la pandemia?, abundan en nuestro día a día. Ahora bien, ¿qué pensarían si les cuento que muchos de los virus que a lo largo de millones de años han infectado a los diferentes seres vivos, aún permanecen, al menos en parte, en su genoma, dispersos entre los genes? La historia a continuación habla de “fósiles” virales que nos acompañan desde hace mucho tiempo.
En busca de “fósiles” virales
Si pensamos en fósiles es probable que venga a nuestra mente la palabra paleontología. En ese sentido, podríamos imaginarnos a una persona en el medio de las montañas, o el lugar que prefieran, caminando atentamente, observando y buscando algún resto fósil para catalogar y analizar, y por medio del mismo, llegar a conocer el pasado de la vida sobre la Tierra.

Pero… cambiemos el contexto. Vayamos a un laboratorio de biología molecular, donde hay personas analizando secuencias de ADN. Ellas también pueden estar buscando “fósiles”, en este caso, moleculares sepultados en el genoma de las especies, es decir, estarán buscando secuencias génicas completas o fragmentos de virus extintos. Estaremos en un laboratorio de paleovirología, un lugar donde se estudian virus antiguos, generalmente en escalas de tiempo prehistóricas o geológicas. Utilizan técnicas genéticas y computacionales para identificarlos o rastrear sus huellas evolutivas en nuestros genomas, es decir, buscan fragmentos de sus secuencias que hayan quedado entre los genes. Lo contaré en detalle.
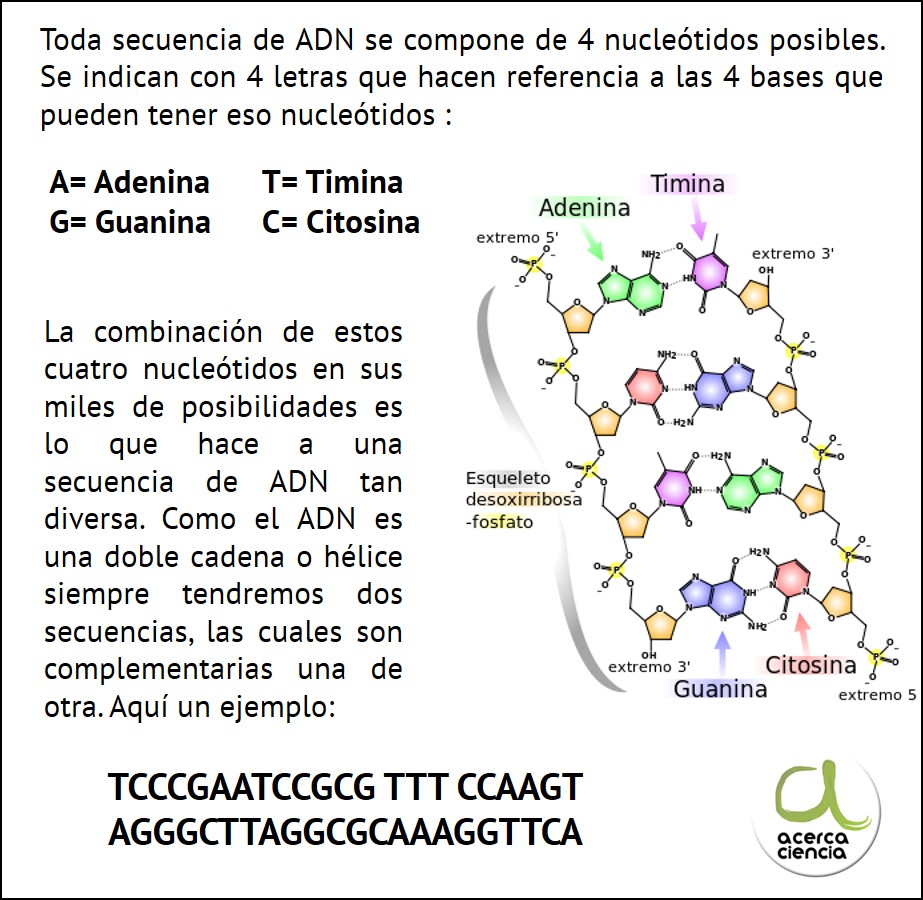
El genoma es la totalidad de la información genética que posee un organismo, y al hablar de los seres humanos en particular, se refiere a la secuencia del ADN (Acido Desoxirribonucleico) contenido en el núcleo de las células somáticas. El genoma refleja cómo las cuatro posibles subunidades, denominadas nucleótidos, están ordenadas en la molécula de ADN. Funcionalmente, proporciona la información necesaria para “construir” el organismo. Se utiliza el término “información” ya que éste no realiza ningún papel activo en la “construcción” del mismo, es solo la secuencia de las subunidades individuales. Más información
¿Qué buscan? ¿Qué son los virus?
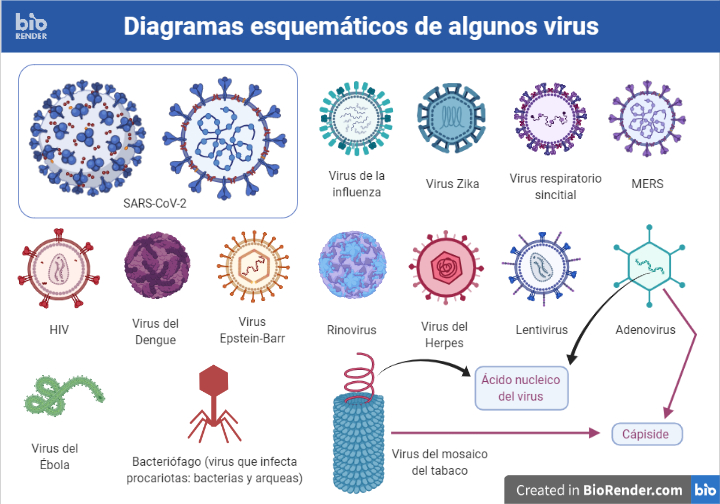
Los virus son agentes infecciosos muy pequeños, de tamaño microscópico, incluso mucho más pequeños que cualquier bacteria. Éstas son hasta 100 veces más grandes que los virus. Constituyen los elementos genéticos más numerosos y diversos de la Tierra. Están en todas partes y son capaces de infectar cualquier tipo de organismo (animales, plantas, algas, hongos, protistas, bacterias y arqueas) así como a otros virus ¡Sí, hay virus que infectan otros virus!
En términos simples, consisten en un centro de ácido nucleico y dependen de un huésped vivo. Actualmente hay un relativo consenso en considerar a los virus como partículas no vivientes porque no están compuestos de células y no pueden realizar actividades metabólicas o reproducirse por cuenta propia.
Básicamente, un virus típico contiene o ácido desoxirribonucleico (ADN) o ácido ribonucleico (ARN), pero no ambos. El ácido nucleico puede ser de una sola cadena o de doble cadena. El centro de ácido nucleico del virus está rodeado por un recubrimiento proteico llamado cápside. Además, algunos virus, llamados virus envueltos, tienen una envoltura membranosa externa que rodea al cápside. La partícula viral completa que está afuera de una célula huésped se llama virión. Éste es la forma en la que el virus se mueve desde la célula donde se produjo hasta una nueva célula huésped que pueda infectar y replicar (hacer una copia) su genoma.
¿Por qué buscar virus «fósiles»?
Los virus han intervenido en la evolución de la vida celular desde su origen ya que pueden transferir sus genes hacia el material genético de sus huéspedes. También pueden transferir genes de un organismo a otro y cruzar las fronteras entre las especies, algo que aprendimos con varios virus, incluido el SARS-Cov-2.
Las recientes técnicas de secuenciación masiva de genomas completos y los análisis bioinformáticos (herramientas computacionales aplicadas a datos biológicos) permiten hoy en día detectar con precisión secuencias de origen viral en los genomas de muchos animales, incluido el humano.
A lo largo de millones de años, estas secuencias han contribuido a la evolución de los genomas de sus huéspedes introduciendo innovación y variabilidad genética e influyen actualmente en la expresión de sus genes. Las personas dedicadas a la paleovirología estudian estos virus extintos, llamados “paleovirus” y los efectos que estos agentes han tenido en la evolución de sus huéspedes.
Escondidos entre nuestros genes
No existe un “registro fósil” físico de los virus; los viriones persisten durante cortos períodos de tiempo y se degradan rápidamente sin dejar rastro directo de su existencia. Pero muchos virus pueden ingresar a los genomas de sus huéspedes y permanecer allí, escondidos entre los genes, latentes, por mucho tiempo. Algunos, como los retrovirus, lo hacen como un paso obligatorio durante su proceso de replicación, y otros pueden hacerlo ocasionalmente, ya sea por accidente o como parte latente de su ciclo de replicación.
Los retrovirus son un tipo de virus con genoma ARN de una sola hebra. Durante su ciclo celular, llevan a cabo la transcripción inversa de su genoma de ARN a ADN, gracias a su enzima viral retrotranscriptasa. ¿Por qué inversa? Porque naturalmente la información genética fluye desde el ADN que se transcribe a ARN mensajero y luego se traduce en proteínas. En los retrovirus ocurre el proceso inverso. Una vez se ha generado la copia de ADN del retrovirus, éste ADN “retrotranscripto” se integra en un cromosoma de la célula huésped pasándose a llamar provirus. Así el retrovirus puede quedar latente insertado en el genoma celular durante mucho tiempo. Un ejemplo típico de un retrovirus es el virus de inmunodeficiencia humana (VIH) que produce el síndrome de inmunodeficiencia adquirida (SIDA). Ciertos virus causantes de cáncer también son retrovirus.
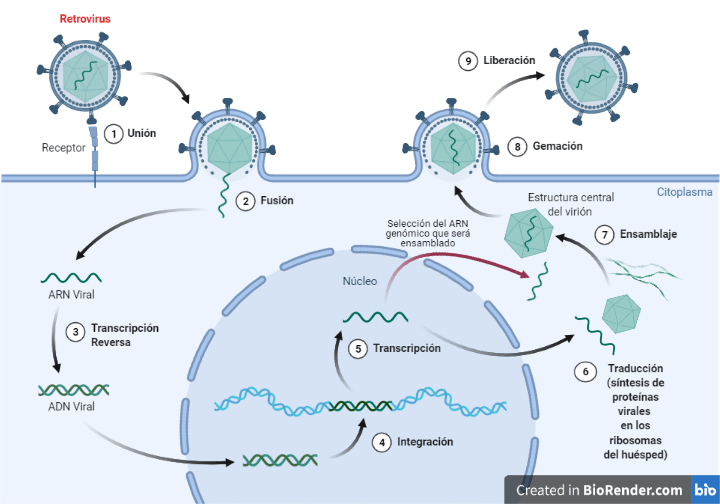
Los retrovirus son los únicos virus conocidos, hasta el momento, que requieren la integración de su genoma en un cromosoma del huésped para completar de forma satisfactoria su ciclo.
Los retrovirus, como todos los virus, pueden transmitirse “horizontalmente” entre distintos individuos y causar infecciones. Pero el virus en forma de provirus, insertado en el genoma de una célula somática del huésped no pasa a la descendencia, no lo heredan sus hijos o hijas.
Sin embargo, ocurre algo distinto si el retrovirus infecta una célula germinal, una célula que dará origen a los gametos, óvulos o espermatozoides. En ese caso, el provirus puede llegar a heredarse como un gen celular más y acabar en el genoma de los descendientes. Si el gameto lleva en su ADN el ADN del virus, después de la fecundación todas las células del nuevo embrión llevarán en su genoma el provirus. Y esto es lo que ha ocurrido en repetidas ocasiones a lo largo de los últimos millones de años en muchos organismos, incluidos los humanos.
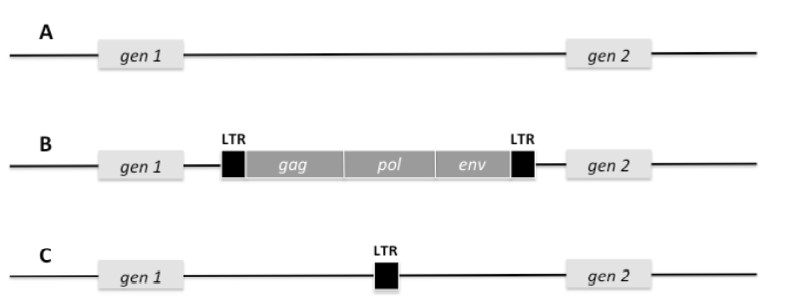
Por eso, en nuestro genoma existen copias de retrovirus que han ido infectando los gametos y se han ido integrando como provirus en el genoma humano durante el curso de la evolución. Son los denominados retrovirus endógenos (ERV, Endogenous RetroVirus; HERV, Human Endogenous RetroVirus), restos de retrovirus que han quedado “fosilizados” en nuestro genoma. En este caso se trata de una transferencia del virus “vertical”, de progenitores a su descendencia, lo que permite la “fijación” de esos ERVs en la población.
Los retrovirus, como todos los virus, pueden transmitirse “horizontalmente” entre distintos individuos y causar infecciones. Pero el virus en forma de provirus, insertado en el genoma de una célula somática del huésped no pasa a la descendencia, no lo heredan sus hijos o hijas.
Sin embargo, ocurre algo distinto si el retrovirus infecta una célula germinal, una célula que dará origen a los gametos, óvulos o espermatozoides. En ese caso, el provirus puede llegar a heredarse como un gen celular más y acabar en el genoma de los descendientes. Si el gameto lleva en su ADN el ADN del virus, después de la fecundación todas las células del nuevo embrión llevarán en su genoma el provirus. Y esto es lo que ha ocurrido en repetidas ocasiones a lo largo de los últimos millones de años en muchos organismos, incluidos los humanos.
Por eso, en nuestro genoma existen copias de retrovirus que han ido infectando los gametos y se han ido integrando como provirus en el genoma humano durante el curso de la evolución. Son los denominados retrovirus endógenos (ERV, Endogenous RetroVirus; HERV, Human Endogenous RetroVirus), restos de retrovirus que han quedado “fosilizados” en nuestro genoma. En este caso se trata de una transferencia del virus “vertical”, de progenitores a su descendencia, lo que permite la “fijación” de esos ERVs en la población.
Si bien no hay consenso completo, los ERV a menudo se clasifican como elementos transponibles (transponsones) y están relacionados con los retrotransposones LTR, es decir, son elementos con la capacidad de transponer o mover secciones de ADN a diferentes posiciones en el genoma.
Recientemente también se reveló que existen representantes endógenos de virus de otros tipos virales, nos solo los retrovirus.
Son parte del “ADN basura”
Nuestro genoma tiene una longitud aproximada de unos 3.200 millones de pares de bases de ADN y contiene unos 20.500 genes. Pero la cantidad no es lo que importa. Un grano de arroz puede llegar a tener el doble de genes que el ser humano. Llamativamente solo el 30% de nuestro ADN codifica genes funcionales. El resto es lo que se denominó durante mucho tiempo y de forma errónea como “ADN basura”, para describir todas las secciones no codificantes de un genoma, la mayoría de las cuales consisten en segmentos repetidos dispersos al azar. Pero hoy se sabe, por ejemplo, que los HERVs forman parte de esos genes no funcionales. Y no son pocos… El 8% de nuestro genoma son HERVs. Tenemos cerca de 450.000 copias de estos retrovirus “fósiles” en nuestro ADN que infectaron hace millones de años nuestra línea germinal. Y lo mismo ha ocurrido en los genomas de muchos otros vertebrados. Debido a su capacidad de copiar su propio ADN e insertarse en otros puntos del genoma, los ERVs se han “extendido” y existen en gran número en el genoma de los mamíferos.
Estos virus o fragmentos de ellos, interfieren en las actividades de la célula huésped
Las secuencias LTR de los extremos de los ERVs de un provirus son secuencias a las que se unen gran cantidad de proteínas de célula con función regulatoria importante. Por eso, estas regiones LTRs pueden interferir con la expresión de otros genes del huésped. Además, los ERVs pueden causar fenómenos de traslocación o reordenamiento de secuencias que están relacionados con procesos cancerígenos.
Los ERVs han contribuido a remodelar la arquitectura del genoma a lo largo de millones de años, y por ello con la evolución del genoma del huésped introduciendo innovación y variabilidad genética, además de influir en la expresión de los genes del huésped.
Algunos ERVs pueden ser patógenos y han sido relacionados con varias enfermedades como esclerosis múltiple, enfermedades autoinmunes, diabetes e incluso cáncer. Cada vez hay más evidencias de que la actividad de varios HERVs contribuye a la formación de tumores, melanomas y carcinomas. Se ha comprobado que las secuencias LTR pueden promover tumores porque generan inestabilidad genética y alteran el patrón de metilación del ADN.
Sin embargo, no todo es negativo. El hecho de que los ERVs se hayan mantenido durante la evolución en nuestro genoma sugiere que han tenido un papel beneficioso para nuestra supervivencia. Hay varios estudios que muestran la implicación de los ERVs en el desarrollo normal de la placenta de los mamíferos. Un ejemplo concreto son los genes de las sincitinas, cuyo origen son en realidad genes env, virales. Las sincitinas son proteínas que promueven la fusión de los trofoblastos, un grupo de células de la capa externa del blastocisto, esenciales para la formación de la placenta de los mamíferos. Algunos ERVs también pueden tener otros efectos beneficiosos: algunos de los productos que codifican sus genes pueden ser responsables de proteger al huésped contra infecciones virales externas.
La caracterización de los ERVs ofrece la oportunidad de llevar a cabo estudios funcionales sobre virus ya extinguidos y sobre otros aspectos de la co-evolución entre los virus y sus huéspedes. Varios grupos de investigación han reconstruido en el laboratorio secuencias completas de ERVs capaces de infectar células. Son versiones infectivas de ERVs similares a los retrovirus antiguos de los que provenían.
En suma, tenemos aún mucho que aprender de estos “fósiles” moleculares.
Bibliografía Consultada
- Eldra Solomon, Linda Berg y Diana Martin. Biología 9ª Edición. 2013. Cengage Learning Editores.
- Aswad A. & Katzourakis, A. Paleovirology: The Study of Endogenous Viral Elements. 2016. Virus Evolution: Current Research and Future Directions Edition: 1 Chapter: 10.
- What is junk DNA, and what is it worth? Scientific American.
- Carlos Sentís. Retrovirus endógenos humanos: Significado biológico e implicaciones evolutivas. Arbor CLXXII, 677 (Mayo), 135-166 pp.
- López-Goñi, I.: Somos lo que somos porque somos virus y bacterias. Nova Acta Científica Compostelana (Bioloxía), 22 (2015).
- Johnson, W., 2019. Origins and evolutionary consequences of ancient endogenous retroviruses. Nature Reviews Microbiology, 17(6), pp.355-370.
- Grandi, N. and Tramontano, E., 2018. Human Endogenous Retroviruses Are Ancient Acquired Elements Still Shaping Innate Immune Responses. Frontiers in Immunology, 9.
- Diehl, W., Patel, N., Halm, K. and Johnson, W., 2016. Tracking interspecies transmission and long-term evolution of an ancient retrovirus using the genomes of modern mammals. eLife, 5.
- Strauss, J. and Strauss, E., 2008. Viruses And Human Disease. 2nd ed. Burlington, MA, Elsevier Academic Press, Chapter 6.
- Eons cannel
Lea el artículo original aquí